Lange Zeit waren WissenschafterInnen nicht in der Lage zu erklären, warum aus einer einzelnen DNA-Sequenz eine Vielzahl von Proteinen hergestellt werden können. Seit wenigen Jahren ist bekannt, dass dies durch Schneiden und Kombinieren der Abschrift dieser DNA geschieht. Diesen Vorgang nennt man „pre-mRNA Spleißen“. Die genaue Erkennung und Definition der Schnittstellen ist von großer biologischer und medizinischer Relevanz: Denn etwa 15 Prozent aller genetischen Erkrankungen werden mit Veränderungen der Schnittstellen in Verbindung gebracht und zahlreiche Krebsarten und Erbkrankheiten sind Abnormalitäten des alternativen Spleißens zuzuschreiben. Die molekularen Details der Erkennung und Definition der Schnittstellen durch Proteine sind größtenteils unbekannt.
Die Arbeitsgruppe um Dr. Tobias Madl vom Institut für Chemie an der Karl-Franzens-Universität Graz konnte in Zusammenarbeit mit der Gruppe um Professor Michael Sattler von der Technischen Universität und Helmholtz Zentrum München erstmals die molekularen Mechanismen aufklären, die grundlegend für die Erkennung, Definition und Regulation der 3‘ RNA Schnittstelle sind. Die Arbeit wurde in der renommierten Fachzeitschrift „Nucleic Acids Research“ als „Featured Article“ publiziert.
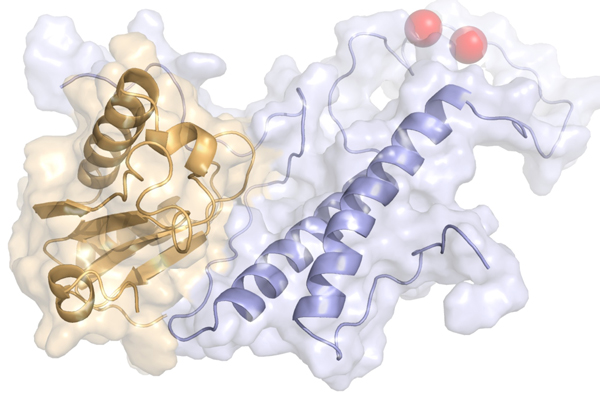
Die ForscherInnen analysierten dafür die dreidimensionale Molekülstruktur der verantwortlichen „molekularen Maschine“ und konnten erstmals zeigen wie die 3‘ RNA Schnittstelle durch einen dynamischen Proteinkomplex, bestehend aus den Proteinen „SF1“ und U2AF65, erkannt und definiert wird. „Das Spleißosom ist eine der kompliziertesten Maschinen innerhalb der Zelle. Es lagert sich ständig um, tauscht Proteine aus und ist überhaupt äußerst dynamisch“, sagt Tobias Madl. Um diese anspruchsvolle Fragestellung überhaupt lösen zu können, verwenden und entwickeln die ForscherInnen seit einigen Jahren innovative Ansätze zur Aufklärung der dreidimensionalen Struktur von Proteinkomplexen. „In Zukunft wird es immer wichtiger die Vorteile verschiedener Techniken der Strukturaufklärung zu kombinieren, um die grundlegenden molekularen Mechanismen von Krankheiten aufklären zu können", erlärt Madl. Die WissenschafterInnen haben dafür bereits Programme entwickelt, die es erlauben, Daten aus Techniken, wie zum Beispiel NMR Spektroskopie, Röntgen/Neutronenkleinwinkelstreuung und Röntgenkristallographie, gleichzeitig zur Strukturrechnung zu verwenden. Sie arbeiten gegenwärtig an der Weiterentwicklungen der Programme zu einer generell anwendbaren Strategie. Solch ein gewaltiger Aufwand, um eine bestimmte Struktur zu erhalten, lohne sich, erklärt Madl. „Auf diese Weise wird der Weg geebnet, zahlreiche Krebsarten und Erbkrankheiten, welche mit Störungen in der Erkennung der 3‘ RNA Schnittstellen in Verbindung gebracht werden, zu verstehen.“
Dr. Tobias Madl forscht als Gastwissenschafter an der Universität Graz sowie als BioSysNet und Emmy Noether Gruppenleiter an der Technischen Universität und am Helmholtz Zentrum München. Seine Forschungsgruppe arbeitet auf dem Gebiet der Strukturbiologie der Signaltransduktion. Die Weiterleitung von Signalen ist essentiell für den Organismus, Störungen darin sind die Ursache einer Vielzahl von Krankheiten (Krebs, Diabetes) und stehen in Verbindung mit Alterung. Ziel der Forschungsgruppe um Madl ist es die grundlegenden molekularen Mechanismen der Wechselwirkungen von Signalproteinen aufzuklären und damit Einblick in die komplizierte Verbindung zwischen deren Funktion und Krankheiten zu erhalten.
Publikation:
“Structure, phosphorylation and U2AF65 binding of the N-terminal domain of splicing factor 1 during 3'-splice site recognition”;
Yun Zhang, Tobias Madl (shared first author & co-corresponding author), Ivona Bagdiul, Thomas Kern, Hyun-Seo Kang, Peijian Zou, Nina Mäusbacher, Stephan A. Sieber, Angela Krämer, Michael Sattler
Nucleic Acids Research, 2013